I.T.C.A
BIOLOGIA MOLECULAR
UNIDAD VII
TRADUCCION DEL ARN MENSAJERO
INTRODUCCION
El ARN mensajero es el acido ribonucleico que contiene
la información genética procedente del ADN ya que este es utilizado en la sintesis de proteinas,
es decir, determina el orden en que se unirán los aminoácidos,
el ARN mensajero es un acido
nucleico monocatenario. Esta
consiste en la síntesis de las
proteínas mediante la unión de aminoácidos según el orden establecido por la
secuencia de nucleótidos del mRNA y el código genético.
OBJETIVOS
Comprender la definición del ARN mensajero
Entender cómo se regula la traducción del ARN
mensajero
Examinar en qué consiste la traducción del ARN
mensajero
7.1 EL CODIGO GENETICO
Es un conjunto de
normas por las que la información codificada en el material genético
(secuencias de ADN o ARN) se traduce en proteínas (secuencias de aminoácidos)
en las células vivas. El código define la relación entre secuencias de tres
nucleótidos, llamadas codones, y aminoácidos. Un codón se corresponde con un
aminoácido específico. El ARN se basa en transportar un mensaje del ADN a la
molécula correspondiente
Las características del código
genético fueron establecidas experimentalmente por Fancis Crick, Sydney Brenner
y colaboradores en 1961.
- El código está organizado en tripletes o codones: Cada tres nucleótidos (triplete) determinan un aminoácido.
- El código genético es degenerado: Existen más tripletes o codones que aminoácidos, de forma que un determinado aminoácido puede estar codificado por más de un triplete.
- El código genético es no solapado o sin superposiciones: Un nucleótido solamente pertenece a un único triplete.
- La lectura es "sin comas": El cuadro de lectura de los tripletes se realiza de forma continua "sin comas" o sin que existan espacios en blanco.
- El código genético nuclear es universal: El mismo triplete en diferentes especies codifica para el mismo aminoácido. La principal excepción a la universalidad es el código genético mitocondrial.
7.2 EL PAPEL DEL ARN EN LA SINTESIS DE PROTEINAS
El ARN, cumple el papel de
determinar el orden en que se uniran los aminoacidos, ya que él, es el
mensajero que lleva la información para la síntesis de proteínas, que tiene
lugar en los ribosomasdel citoplasma celular. Los aminoácidos son transportados
por el ARN de transferencia (ARNt) , específico para cada uno de ellos, y son llevados
hasta el ARN mensajero (ARNm), dónde se aparean el codón de éste y el
anticodóndel ARN d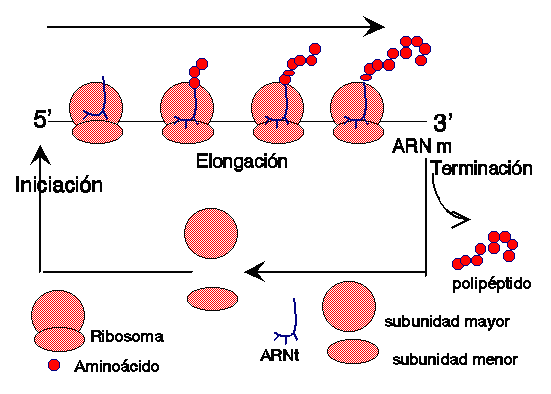 e transferencia, por complementariedad de bases, y de ésta
forma se sitúan en la posición que les corresponde.
e transferencia, por complementariedad de bases, y de ésta
forma se sitúan en la posición que les corresponde.
Una vez finalizada la síntesis de una proteína, el ARN mensajero queda libre y puede ser leído de nuevo.
 e transferencia, por complementariedad de bases, y de ésta
forma se sitúan en la posición que les corresponde.
e transferencia, por complementariedad de bases, y de ésta
forma se sitúan en la posición que les corresponde. Una vez finalizada la síntesis de una proteína, el ARN mensajero queda libre y puede ser leído de nuevo.
*El ARN mensajero (ARNm o RNAm) lleva la
información sobre la secuencia de aminoácidos de la proteína desde el ADN,
lugar en que está inscrita, hasta el ribosoma, lugar en que se sintetizan las
proteínas de la célula. Es, por tanto, una molécula intermediaria entre el ADN
y la proteína y el apelativo de "mensajero" es del todo
descriptivo.
*El
ARN mensajero (ARNm o RNAm) lleva la información sobre la secuencia de
aminoácidos de la proteína desde el ADN, lugar en que está inscrita, hasta el
ribosoma, lugar en que se sintetizan las proteínas de la célula. Es, por tanto,
una molécula intermediaria entre el ADN y la proteína y el apelativo de
"mensajero" es del todo descriptivo.
*ARN de transferencia: Son cortos polímeros de unos 80 nucleótidos que
transfiere un aminoácido específico al polipéptido en crecimiento; se unen a
lugares específicos del ribosoma durante la traducción. Tienen un sitio
específico para la fijación del aminoácido (extremo 3') y un anticodón formado
por un triplete de nucleótidos que se une al codón complementario del ARNm
mediante puentes de hidrógeno.
*ARN ribosómico. El ARN ribosómico (ARNr o RNAr) se halla combinado con proteínas para formar los ribosomas, donde representa unas 2/3 partes de los mismos
*ARN ribosómico. El ARN ribosómico (ARNr o RNAr) se halla combinado con proteínas para formar los ribosomas, donde representa unas 2/3 partes de los mismos
7.2.1 TIPOS DE ARN
El ARN es la molécula que usan las células para
poder convertir la información genética que está en el ADN a proteínas. Para
hacer esto el ARN pasa por varias fases:

ARN ribosómico: es el que se encarga de hacer las proteínas formando parte de los ribosomas.
ARN mensajero: este es el ARN que conseguimos de la cadena de ADN. Determina el orden de los aminoácidos que debe tener la proteina que queremos crear.
ARN de transferencia: se encarga de llevar los aminoácidos necesarios para crear la proteína codificada por el ARN mensajero al ribosoma
ARN de interferencia: es un método de regulación genética ya que suprime expresión de genes mediante métodos de ribointerferencia (conducen a la degradación del ARN mensajero, del que son complemntarias)
ARN nucleolar: es una molécula pequeña que está y se sintetiza en el nucleolo de las celulas eucariotas a partir de la transcripción de ADN. Es el precursor e indispensable para la síntesis de parte del ARN ribosómico.

ARN ribosómico: es el que se encarga de hacer las proteínas formando parte de los ribosomas.
ARN mensajero: este es el ARN que conseguimos de la cadena de ADN. Determina el orden de los aminoácidos que debe tener la proteina que queremos crear.
ARN de transferencia: se encarga de llevar los aminoácidos necesarios para crear la proteína codificada por el ARN mensajero al ribosoma
ARN de interferencia: es un método de regulación genética ya que suprime expresión de genes mediante métodos de ribointerferencia (conducen a la degradación del ARN mensajero, del que son complemntarias)
ARN nucleolar: es una molécula pequeña que está y se sintetiza en el nucleolo de las celulas eucariotas a partir de la transcripción de ADN. Es el precursor e indispensable para la síntesis de parte del ARN ribosómico.
7.2.2 ESTRUCTURA RIBOSOMAL
Los ribosomas son grandes complejos
ribonucleoproteicos en los que se sintetizan las proteínas. En este proceso,
los codones del RNA mensajero (mRNA) son leídos por los anticodones de los RNA
de transferen cia (tRNA), las moléculas adaptadoras que portan aminoácidos
específicos para esos codones. Estos aminoácidos se añaden a una cadena de
proteína en crecimiento, formándose los enlaces peptídicos en el interior del
ribosoma.
cia (tRNA), las moléculas adaptadoras que portan aminoácidos
específicos para esos codones. Estos aminoácidos se añaden a una cadena de
proteína en crecimiento, formándose los enlaces peptídicos en el interior del
ribosoma.
El ribosoma 70S está formado por dos subunidades:
una mayor, 50S, y otra menor, 30S. La subunidad 50S contiene un rRNA 23S y otro
5S, y más de 30 proteínas, 22 de las cuales se han podido resolver en la
estructura del cristal. La subunidad 30S contiene un rRNA 16S y 20 proteínas.
Los RNA de cada subunidad forman el núcleo estructural y funcional del
ribosoma. Las proteínas ribosomales están implicadas en puentes entre las
subunidades y en contactos con los tRNA, y apoyan la función de los RNA de cada
subunidad. Se puede ver la posición y conformación de los rRNA componentes de
cada subunidad de este modo:
- El rRNA 16S de la subunidad menor, 30S, se pliega formando cuatro dominios: 5', central, 3' principal y 3' secundario. Estos dominios tienen autonomía estructural, lo que significa que se mueven unos respecto a otros durante la síntesis de proteínas. Visto desde la interfaz entre subunidades, el rRNA 16S forma la mayor parte de la superficie de contacto, mientras que las proteínas se sitúan principalmente en la periferia.
- El rRNA 23S de la subunidad mayor, 50S, se pliega formando seis dominios estructurales secundarios que contienen más de 130 hélices de RNA: dominios I, II, III, IV, V, VI. Estos seis dominios, a diferencia de los del rRNA 16S en la subunidad menor, están profundamente entrelazados. El rRNA 5S forma un séptimo dominio de estructura terciaria en la subunidad mayor. Al igual que el rRNA 16S de la subunidad menor, los rRNA de la subunidad mayor forman la mayor parte de la superficie de contacto entre subunidades, mientras que las proteínas se sitúan principalmente en la periferia.
7.2.3 PROCARIOTICO
En una célula procariota suelen existir entre 10.000 y 15.000 ribosomas,
orgánulo portador de la mayoría de las actividades enzimáticas implicadas en la
biosíntesis. Contienen el sitio A o aceptor o aminoacilo, donde entr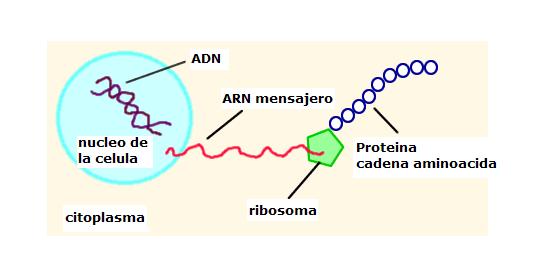 a el
aminoacil-tRNA. El sitio P, peptidil o donador, es donde está el
peptidil-tRNA. También está el sitio E o de eyección o salida que está
ocupado por el tRNA que ya no porta aminoácido. En la parte superior de la
subunidad mayor, cerca del enlace que une los tRNA con su péptido y su aminoácido,
está el sitio peptidiltransferasa. Recordemos que tanto el sitio P como
el A se forman correctamente cuando se unen ambas subunidades. Sin embargo, el
sitio E y el peptidil-transferasa tienen todos sus componentes en la subunidad
mayor.
a el
aminoacil-tRNA. El sitio P, peptidil o donador, es donde está el
peptidil-tRNA. También está el sitio E o de eyección o salida que está
ocupado por el tRNA que ya no porta aminoácido. En la parte superior de la
subunidad mayor, cerca del enlace que une los tRNA con su péptido y su aminoácido,
está el sitio peptidiltransferasa. Recordemos que tanto el sitio P como
el A se forman correctamente cuando se unen ambas subunidades. Sin embargo, el
sitio E y el peptidil-transferasa tienen todos sus componentes en la subunidad
mayor.
 a el
aminoacil-tRNA. El sitio P, peptidil o donador, es donde está el
peptidil-tRNA. También está el sitio E o de eyección o salida que está
ocupado por el tRNA que ya no porta aminoácido. En la parte superior de la
subunidad mayor, cerca del enlace que une los tRNA con su péptido y su aminoácido,
está el sitio peptidiltransferasa. Recordemos que tanto el sitio P como
el A se forman correctamente cuando se unen ambas subunidades. Sin embargo, el
sitio E y el peptidil-transferasa tienen todos sus componentes en la subunidad
mayor.
a el
aminoacil-tRNA. El sitio P, peptidil o donador, es donde está el
peptidil-tRNA. También está el sitio E o de eyección o salida que está
ocupado por el tRNA que ya no porta aminoácido. En la parte superior de la
subunidad mayor, cerca del enlace que une los tRNA con su péptido y su aminoácido,
está el sitio peptidiltransferasa. Recordemos que tanto el sitio P como
el A se forman correctamente cuando se unen ambas subunidades. Sin embargo, el
sitio E y el peptidil-transferasa tienen todos sus componentes en la subunidad
mayor.
7.2.4 EUCARIOTICO
La estructura eucariotas es
básicamente el mismo que el de procariotas, con las diferencias principales:
- El ribosoma y sus subunidades son más grandes (40S + 60 S → 80S).
- Los rRNA son mayores y hay más proteínas por subunidad ribosómica.
- La subunidad mayor contiene los rRNA 28S y 5S, pero además una 5,8S adicional que no existe en los procariotas.
- El ribosoma no tiene sitio E.
7.3 ETAPAS DE LA SINTESIS DE PROTEINAS EN ORGANISMOS PROCARIOTICOS
|
Se comienza con la subunidad menor sola. IF-1 se
une a la base del sitio A para forzar que el primer fMet-tRNA entre en el
sitio P.
IF-3 se le necesita para estabilizar la subunidad 30S
IF-2 sirve para depositar el aminoacil-tRNA (fMet-tRNA en este caso)
en el ribosoma.
Los 3 IF junto con el mRNA, el fMet-tRNA y la
subunidad 30S forman el complejo de iniciación
|
El tRNA iniciador que reconoce
el AUG (en ocasiones GUG y raramente UUG) es especial ya que porta una
formil-Met, presenta modificaciones postranscripcionales específicas, sólo
puede usarse en iniciación, y es el único capaz de entrar en el sitio P (no el
A) sin la subunidad mayor del ribosoma.
ELONGACIÓN
El crecimiento de la cadena polipeptídica en el
ribosoma es un proceso cíclico que se repite tantas veces como aminoácidos se
incorporen. Cada ciclo consta de 4 pasos: ubicación del nuevo aa-tRNA,
verificación o corrección del aminoácido introducido, formación del enlace
peptídico y translocación. Los sitios E y A cooperan negativamente puesto que
nunca que encuentran ocupados a la vez.
Corrección
Para dejar el aa-tRNA en su sitio, EF-Tu tiene que
hidrolizar el GTP. Esto es un proceso relativamente lento, que da tiempo a
verificar el apareamiento codón-anticodón. Si el apareamiento codón-anticodón
es incorrecto, el aminoacil-tRNA se rechaza y queda de nuevo libre el sitio A
para aceptar el aminoacil-tRNA correcto.
Transpeptidación
La cadena polipeptídica enganchada al tRNA del
sitio P se transfiere sobre el aminoácido transportado por el tRNA del sitio A.
Esta transferencia la cataliza el sitio peptidil transferasa de la
subunidad 50S. Concretamente, el rRNA 23S alojado en este sitio catalítico es
quien realiza la función catalítica fundamental, actuando como ribozima.
Translocación
(traslado)
El tRNA descargado del sitio P se transfiere
al E y el tRNA que tiene el péptido en el sitio A pasa al P. El desplazamiento
hace que el ribosoma avance 3 nt por el mRNA.
Tras la translocación, la
cooperación negativa entre E y A hace que no pueda entrar otro aa-tRNA nuevo en
A hasta que el que hay en E no ha salido. En este momento se ha completado el
ciclo, con la diferencia de que ahora la cadena polipeptídica ha crecido en un
residuo y el ribosoma está desplazado 3 nt en el mRNA.
TERMINACION
Determina
la conclusión de la síntesis de la proteína cuando el sitio A del ribosoma es
abordado por el codón de terminación
del ARNm (UUA, UGA o UAG,
indistintamente). Ello deja al sitio A sin el esperado aminoacil-ARNtAA,
aunque pronto es ocupado por un factor
de terminación llamado eRF (eucaryotic releasing factor), que sabe
reconocer a los tres codones de terminación.
En
síntesis la terminación de la cadena polipeptídica está señalada por el ARNm
mediante un codón que no especifica la incorporación de ningún aminoácido . Ese
codón de terminación puede ser UUA, UGA o UAG, y sobre él no se une ningún
ARNt. En cambio, es reconocido por dos proteínas llamadas factores de
liberación (eRF). Cuando esto sucede, la proteína terminada se libera del
último ARNt, que también se separa del ARNm. Por último también se disocian las
subunidades ribosómicas. Todos estos
elementos pueden ser reutilizados en una
nueva síntesis.
7.4 ETAPAS DE LA SINTESIS
DE PROTEINAS EN ORGANISMO EUCARIOTICOS
INICIO
EI F-6 esta biliza la subunidad 60S del ribosoma. eIF-3, eIF-1 y
eIF-1A se unen a la subunidad menor. Junto con el  aa-tRNA unido a eIF-2,
van a formar el complejo 43S. Como en procariotas, los aa-tRNA llegan
acompañados de un factor (eIF-2) que se reciclará mediante el factor eIF-2B.
Gracias a eIF-5B, el Met-tRNAi se coloca correctamente.
aa-tRNA unido a eIF-2,
van a formar el complejo 43S. Como en procariotas, los aa-tRNA llegan
acompañados de un factor (eIF-2) que se reciclará mediante el factor eIF-2B.
Gracias a eIF-5B, el Met-tRNAi se coloca correctamente.
ELONGACION
Los factores de elongación que
intervienen en eucariotas son análogos a los procariotas:
·  eEF-1α
que es una proteína G, análogo a EF-Tu (EF-1A) [el que aporta los
aminoacil-tRNA al ribosoma]. Reconoce cualquier tRNA menos el iniciador. Como
no tiene afinidad por el ribosoma, se desprende de él cuando el aa-tRNA se fija
al ribosoma con hidrólisis de GTP.
eEF-1α
que es una proteína G, análogo a EF-Tu (EF-1A) [el que aporta los
aminoacil-tRNA al ribosoma]. Reconoce cualquier tRNA menos el iniciador. Como
no tiene afinidad por el ribosoma, se desprende de él cuando el aa-tRNA se fija
al ribosoma con hidrólisis de GTP.
·
eEF-1βγ
que es análogo a EF-Ts (EF-1B) [su función es reciclar y regenerar el eEF-1α],
·
eEF-2 es
otra proteína G análoga a EF-G (EF-2) que interviene en la translocación del
ribosoma.
Se une al ribosoma en el
sitio A cuando aparece cualquier codón de terminación, alterando las
propiedades  hidrofóbicas del sitio peptidil-transferasa. También tiene el motivo
GGQ que altera la actividad del sitio peptidil-transferasa para que ahora
sea el agua quien actúe como agente nucleofílico. El tRNA está en el sitio P
del ribosoma y el eRF1 en el sitio A. Para disociar este complejo y regerenar
el ribosoma útil, entra en funcionamiento el factor eRF3 —otra proteína
G que se parece a eEF-1α— que ha estado en todo momento físicamente asociado a
eRF1 (por eso antes se creía que sólo había un factor, el eRF). eRF3 lleva una
molécula de GTP que se hidroliza para permitir esta liberación.
hidrofóbicas del sitio peptidil-transferasa. También tiene el motivo
GGQ que altera la actividad del sitio peptidil-transferasa para que ahora
sea el agua quien actúe como agente nucleofílico. El tRNA está en el sitio P
del ribosoma y el eRF1 en el sitio A. Para disociar este complejo y regerenar
el ribosoma útil, entra en funcionamiento el factor eRF3 —otra proteína
G que se parece a eEF-1α— que ha estado en todo momento físicamente asociado a
eRF1 (por eso antes se creía que sólo había un factor, el eRF). eRF3 lleva una
molécula de GTP que se hidroliza para permitir esta liberación.
7.4.1 MODIFICASION DE
PROTEINAS POSTRADUCCION
Conjunto
de procesos que modifican las proteínas una vez ha terminado su síntesis, con
el objetivo de contribuir a su correcto plegamiento, a su activación o a la
regulación de su actividad o situación en el interior de la célula. Se han
descrito más de 100 modificaciones post - traduccionales.
Las modificaciones post – traduccionales no son
meras decoraciones de los aminoácidos sino que determinan: actividad de la
proteína, localización, recambio o degradación e interacciones con otras
proteínas
La actividad de las proteínas no está únicamente controlada por la velocidad de síntesis y degradación sino que también por procesos específicos y selectivos de modificación covalente o modificación post-traduccional que modula interacciones moleculares, localización de proteínas y estabilidad.
La actividad de las proteínas no está únicamente controlada por la velocidad de síntesis y degradación sino que también por procesos específicos y selectivos de modificación covalente o modificación post-traduccional que modula interacciones moleculares, localización de proteínas y estabilidad.



No hay comentarios:
Publicar un comentario