
 INSTITUTO
TECNOLOGICO
INSTITUTO
TECNOLOGICO
DE CD ALTAMIRANO
BIOLOGIA MOLECULAR
ALUMNA
MARICRUZ HERRERA MAYA
PROFESOR
FRANCISCO JAVIER PUCHE ACOSTA
CARRERA: LIC.BIOLOGIA
SEMESTRE: VI GRUPO: "A"
UNIDAD 4
REPLICACION DEL ADN
INTRODUCCION
El ácido desoxirribonucleico, frecuentemente abreviado como ADN. Hasta
hace unos años era completamente desconocido para todos como era que
funcionaba la sangre y sobre todo de que manera podían pasar las
características de un ser vivo a otro este descubrimiento hecho por
james Watson y Crick aun son importantes para nuestras vidas
OBJETIVOS
* Conocer la historia del descubrimiento del ADN, para que sirva, como
funciona, de qué manera afecta o ayuda a los científicos e identificar
cómo funcionan las bases nitrogenadas en el ADN y en los seres vivos.
* Describir el proceso de la replicasion del ADN.
4.1 REPLICACION DEL GENOMA PROCARIOTE

En este proceso interviene
un grupo de enzimas conocido genéricamente como ADN polimerasa. Este complejo
multienzimático va incorporando nucleótidos a la cadena que se va sintetizando
utilizando como molde una de las hebras de la cadena antigua. Esta polimerización se
produce añadiendo nuevos nucleótidos al extremo 3' de una hebra de ADN en crecimiento.
Por ello se dice que la síntesis del ADN se produce en dirección 5'<3' mientras la
ADN polimerasa va leyendo la hebra molde en dirección 3'o5'.
Por consiguiente, para que se produzca la síntesis de ADN por la ADN
polimerasa, además de la enzima que realiza la polimerización en sí, es necesario otro
juego de proteínas que van desenrollando la doble hélice de ADN delante de la ADN
polimerasa para permitir que ésta funcione. Estas enzimas se denominan genéticamente
topoisomerasas y girasas. La acción de las topoisomerasas y girasas permite abrir unas
burbujas en la doble hélice de ADN en las que entra el complejo de la ADN polimerasa y
realiza la polimerización.

Sin embargo, la ADN polimerasa no es capaz de iniciar la
polimerización del ADN sino que solamente es capaz de continuarla. Para que la ADN
polimerasa funcione es necesario que exista un extremo 3' libre al que añadir un nuevo
nucleótido. ¿Quién pone ese extremo 3' libre?. En todos los casos (salvo algunas
excepciones de ciertos virus) ése extremo 3' viene de una pequeña cadena de ARN que se
ha sintetizado como paso inicial de la replicación del ADN. Por consiguiente, la
replicación del ADN comienza con la síntesis de una pequeña molécula de ARN que luego
continúa como molécula de ADN. Esta pequeña molécula de ARN se denomina cebador o
primer„ y es sintetizada por otro complejo multienzimático denominado ARN
polimerasa.
La síntesis de ADN (replicación) va procediendo detrás de la
horquilla que se forma en el punto en que se van abriendo las dos hebras de ADN molde por
acción del complejo de girasas y topoisomerasas. De esta forma, la hebra que se va
sintetizando es continua. Sin embargo, la hebra que se sintetiza usando como molde la
complementaria de la anterior no puede ser continua porque la acción de las
topoisomerasas y girasas se produce detrás del punto de origen de la transcripción. Por
esto, de las dos hebras hijas, una será continua y l a otra discontinua. Los fragmentos
de ésta última se unirán entre sí mediante la acción de otra enzima denominada ADN
ligasa que une los fragmentos anteriores.
En las bacterias, cuyo material genético es cerrado, no tiene
extremos, sólo existe un origen de replicación del ADN en cada molécula de ADN. Esto es
válido para los plásmidos y para los cromosomas bacterianos. Asimismo, esto se cumple
también en el ADN de los orgánulos celulares procarióticos presentes en las células
eucarióticas.
 En las células procariotas
En las células procariotas
Hay 1 lugar de origen de replicación que se muestra en
la horquilla de replicación (replication fork) y que señala
el avance de la copia. La horquilla (fork) indica que se está
haciendo la separación y la replicación a la vez.
El avance es bidireccional, lo que acorta el tiempo. En el sitio
en que empieza la replicación se organizan las proteínas
en un complejo llamado replisma. La replicación del ADN
en procariotas sucede a una velocidad de 500 nucleótidos
por segundo.
4.2 REPLICACION DEL GENOMA EUCARIOTE
La replicación del ADN
eucariótico es esencialmente igual a la del procariótico aunque presenta ciertas
diferencias los cromosomas eucarióticos son abiertos, en lugar de cerrados, y el número
de orígenes de transcripción es múltiple en cada cromosoma en lugar de tener uno solo
por cromosoma.
En las células procariotas
El proceso es esencialmente el mismo pero el ADN es mucho más
grADNe y linear. Hay varios orígens de replicación
y es bidireccional. El avance es más lento que en procariotas
ya que hay más proteínas asociadas al ADN que hay
que soltar. La replicación del ADN, que ocurre una sola
vez en cada genración celular necesita de muchos "ladrillos",
enzimas y una gran cantidad de energía en forma de ATP
(recuerde que luego de la fase S del ciclo celular , las células
pasan a una fase G a fin de, entre otras cosas, recuperar energía
para la siguiente fase de la división celular). La replicación
del ADN en el ser humano se realiza a una velocidad de 50 nucleótidos
por segundo. Los nucleótidos tienen que ser armados y
estar disponibles en el núcleo conjuntamente con la energía
para unirlos.
Prácticamente la mitad del ADN eucariota corresponde a secuencias nucleotídicas
repetidas.
|
Existen secuencias altamente repetitivas (de función
desconocida, salvo para la secuenciación génica del hombre):
|
* SATÉLITES: 5-50 pares de bases, repetidas hasta un
millón de veces.
|
|
* MINISATÉLITES: 12-100 pares de bases, repetidas un
número variable de veces, son marcadores genéticos moleculares de cada
persona.
|
|
* MICROSATÉLITES: 1-5 pares de bases, en grupos de 10-50
copias.
|
|
Secuencias moderadamente repetitivas:
|
* Telómeros, se encuentran en los extremos de los
cromosomas. En el ser humano esta secuencia es TTAGGG y se repite unas
2500 veces. Recordando la replicación del ADN... la cadena avanzada
puede replicarse hasta el extremo, pero como la cadena retrasada no
tiene un cebador en el extremo 5´ del cromosoma, en cada replicación
se produce un acortamiento de la nueva hebra formada. De este modo el
nuevo ADN carece de esta última porción repetitiva ya que el mecanismo
de reparación corta este extremo. En células en activa división
(médula ósea y línea germinal) una telomerasa cataliza el agregado de
la porción faltante.
|
|
* Secuencias codificadores de ARNt y ARNr: existen
múltiples copias de ADN que codifican para estos ARN, ya que se precisan
en grandes cantidades
|
4.3 CONTROL GENETICO DE LA REPLICACION
Para
que la replicación ocurra, la célula necesita las moléculas que
constituyen los diferentes nucleótidos y una serie de enzimas que
controlan el proceso en todo momento. Así es como se realiza:
1.-
La replicación comienza con la rotura de los enlaces que unen las bases
nitrogenadas complementarias de ambas cadenas. De esta forma, las dos
hebras comienzan a desenrollarse. En este proceso intervienen enzimas
que permiten la separación.
2.-
Cada cadena va a servir de molde para la síntesis de una nueva cadena
complementaria. Cuando ambas se separan, enzimas específicas van leyendo
la información y uniendo los nucleótidos complementarios.
3.-
De esta manera quedan formadas dos nuevas cadenas dobles. Ahora, cada
una de las nuevas cadenas de doble hélice contiene una de las hebras del
ADN de origen y otra nueva, por eso se dice que la replicación es semiconservativa.
El
tiempo que tarda el ADN en replicarse es de alrededor de siete horas.
En realidad, semejante proceso tendría que durar mucho más si la
duplicación se realizara de una punta a la otra de cada cromosoma. Sin
embargo, esto no ocurre. La célula utiliza “estrategias” mediante las
cuales el material genético se duplica en períodos más cortos. La
replicación del ADN ocurre en varios puntos a la vez, en distintos
tramos de la molécula.
A.-
Las enzimas separan las dos hebras de la cadena helicoidal de ADN en
puntos específicos. Las enzimas ADN helicasa desenrollan cada hebra de
ADN.
B.- La enzima ADN polimerasa
lee cada hebra y fabrica una nueva uniendo los nucleótidos
complementarios. La lectura de la cadena molde avanza hacia un lado en
una hebra y hacia el otro, en la otra.
C.- Finalmente, se obtienen dos cadenas nuevas de ADN, una por cada hebra del ADN original.
•Si
lleva la información genética es necesario que se duplique para poder
repartirla a las células hijas.
•Desde
principios del S. XX, se sabe que los cromosomas llevan la información
genética, ¿cómo se sabe?:
–El
espermatozoide sólo pasa su núcleo al óvulo, en el núcleo están los cromosomas.
–Si
los cromosomas están formados por proteínas y ADN , ¿quién de los dos lleva la
información genética?
•Una experiencia que ayudó a resolver esta
incógnita, fue la experiencia realizada por Griffith (1928) y explicada por
Avery, MacLeod y McCarthy en 1952:
Explicación: Las bacterias R, se transforman en S y
producen la muerte del ratón. ¿Cómo?. Al calentar se destruyen las proteínas
pero no el
ADN
por lo que fragmentos de ADN de las S pueden entrar en las R y las
transforman en S el ADN lleva
la información genética.
Primeras hipótesis sobre la replicacion del ADN
·
Semiconservativa
ConservativaDispersiva
4.4
REPLICACION IN VITRO DEL ADN (PCR)
 Kornberg (discípulo de Ochoa), en 1956,
aisló la enzima ADN-polimerasa (enzima capaz de unir nucleótidos de ADN in
vitro), realizó la siguiente experiencia:
Kornberg (discípulo de Ochoa), en 1956,
aisló la enzima ADN-polimerasa (enzima capaz de unir nucleótidos de ADN in
vitro), realizó la siguiente experiencia:
ADN polimerasa + Mg +2 +
nucleótidos
trifosfato + hebra patrón
no se unían nucleótidos complementarios.
- ADN polimerasa + Mg +2
+ nucleótidos trifosfato + hebra patrón + un cebador (fragmento
complementario)
se unían nucleótidos complementarios a la hebra patrón, pero siempre siguiendo
la dirección de crecimiento 5’ 3’.
Reacción
en Cadena de la Polimerasa (PCR) Es una técnica clave utilizada en la genética
molecular para reproducir una secuencia relativamente corta de ADN; este segmento
de ADN a menudo es un gen o una parte crítica del gen y amplifica por PCR (Es decir, hace más copias de) la secuencia de interés que nos permite estudiar lo
que la secuencia particular. Antes de
PCR fue inventada, la amplificación de ADN se llevó a cabo en bacterias y tomó
semanas para llevar a cabo. PCR
utiliza las mismas moléculas que la naturaleza utiliza para copiar el ADN.
Con el fin de determinar si está o no amplificado
el gen correcto o para comprobar si ese gen está presente, se utiliza electroforesis
de agarosa. Agarosa crea una matriz que
permite que las pequeñas moléculas de ADN para viajar más rápido y más grandes
moléculas de ADN para viajar más lento separando así las moléculas de ADN de
acuerdo con el tamaño cuando se aplica a través de un campo eléctrico.
Explicación
A. Las reacciones de ciclismo:
Hay tres pasos principales en una
PCR, que se repiten durante 30 o 40 ciclos. Esto se hace en un ciclador térmico automatizado, que puede calentar y
enfriar los tubos con la mezcla de reacción en un tiempo muy corto.
- Desnaturalización (94-95 ° C) :
Durante la desnaturalización, la doble cadena se funde, o desnaturaliza (Lo que significa abre) , Con el ADN de cadena
sencilla y todas las reacciones enzimáticas detener (Por ejemplo: la extensión
de un ciclo anterior) .
- Recocido (54-60 ° C) :
Los cebadores se jiggling alrededor de la cual es causada por el
movimiento browniano. Los enlaces de hidrógeno se forman
constantemente y roto entre el cebador de cadena sencilla y la plantilla
monocatenario. Los bonos más estables
duran un poco más (Cebadores que se ajustan
exactamente) y en ese pequeño pedazo de
ADN de doble cadena (Plantilla y cartilla) , La polimerasa puede
conectar y empezar a copiar la plantilla. Una vez que hay unas pocas
bases construidas en los enlaces de hidrógeno son tan fuertes entre la
plantilla y la cartilla de que no se rompe más. La temperatura real de
recocido está determinada por la secuencia del cebador utilizando sus
temperaturas de fusión, referidos como Tm.
- Extensión (68 - 74 ° C) :
La extensión es cuando los nucleótidos libres que complementan la sola
hebra de ADN se añadió en el extremo 3 'del cebador. Esta es la temperatura de
trabajo ideal para la polimerasa. Los cebadores tienen una atracción fuerte a
la plantilla, creado por enlaces de hidrógeno. Es más difícil de romper
estos lazos de lo que es para añadir nucleótidos libres a los cebadores. Los cebadores pueden unirse
al ADN monocatenario incluso si no son exactamente complementaria. Pero si el
"partido" está con muy pocos nucleótidos, entonces hay un menor
número de enlaces de hidrógeno. Así, el cebador puede caerse el nucleótido
monocatenario debido a la temperatura más alta y no habrá extensión del
fragmento (La única hebra de ADN que
está intentando copiar.)
Las bases (Complementaria a la
plantilla) están acoplados a la
imprimación sobre el lado 3 ' (La polimerasa añade dNTP desde 5 'a 3', la
lectura de la plantilla de 3 'a 5' lado, las bases complementarias se
añaden a la plantilla)
Problema de la dirección en la replicacion del ADN in vivo
Cairns en 1963:
E. coli en un medio con timina marcada con tritio
(H3), que emite partículas β que son captadas por película fotográfica.
*
Cada 2’ ó 3’ depositaba el cultivo
sobre la placa fotográfica.
4.4.1 SECUENCIAS DEL ADN
Las posibles letras son A, C, G,
y T, que simbolizan las cuatro subunidades de nucleótidos de una banda ADN - adenina, citosina, guanina, timina, que son
bases covalentemente ligadas a cadenas fosfóricas. En el típico caso, las
secuencias se presentan pegadas unas a las otras, sin espacios, como en la
secuencia AAAGTCTGAC, yendo de 5' a 3' de izq. a derecha.
Una sucesión de cualquier número de nucleótidos
mayor a cuatro es pasible de llamarse una secuencia. En relación a su función
biológica, que puede depender del contexto, una secuencia puede tener sentido o antisentido,
y ser tanto codificante o no codificante. Las secuencias de ADN pueden
contener "ADN no codificante."
Las secuencias pueden derivarse de material
biológico de descarte a través del proceso de secuenciación de ADN.
En algunos casos especiales, las letras seguidas de
A, T, C, y G se presentan en una secuencia. Esas letras representan ambiguedad.
De todas las moléculas muestreadas, hay más de una clase de nucleótidos en esa
posición. Las reglas de la Unión Internacional de Química
Pura y Aplicada (IUPAC) son las
que siguen:
A = adenina
C = citosina
G = guanina
T = timina
R = G A (purina)
Y = T C (pirimidina)
K = G T (keto)
M = A C (amino)
S = G C (enlaces fuertes)
W = A T (enlaces débiles)
B = G T C (todos y A)
D = G A T (todos y C)
H = A C T (todos y G)
V = G C A (todos y T)
N = A G C T (cualquiera)
Secuenciación de Maxam-Gilbert
 En 1976-1977, Allan Maxam y Walter Gilbert desarrollaron un método para secuenciar ADN basado
en la modificación química del ADN y posterior escisión en bases específicas
Aunque Maxam y Gilber publicaron su secuenciación química dos años antes del
trascendental artículo de Sanger y Coulson sobre su método de secuenciación
"más-menos",la secuenciación de Maxam y Gilbert rápidamente se hizo
más popular hasta que se pudo utilizar ADN directamente, mientras que el método
inicial de Sanger requería que cada comienzo de lectura fuera clonado para
producir un ADN de cadena simple. No obstante, con el desarrollo y mejora del
método de terminación de la cadena (ver más adelante), la secuenciación de
Maxam y Gilbert ha quedado en desuso debido a su complejidad técnica, el uso
extensivo de productos químicos peligrosos y dificultades para escalarla.
Además, a diferencia del método de terminación de la cadena, los reactivos que
se usan en el método de Maxam y Gilbert no se pueden adaptar para utilizarse en
un kit biológico estándar.
En 1976-1977, Allan Maxam y Walter Gilbert desarrollaron un método para secuenciar ADN basado
en la modificación química del ADN y posterior escisión en bases específicas
Aunque Maxam y Gilber publicaron su secuenciación química dos años antes del
trascendental artículo de Sanger y Coulson sobre su método de secuenciación
"más-menos",la secuenciación de Maxam y Gilbert rápidamente se hizo
más popular hasta que se pudo utilizar ADN directamente, mientras que el método
inicial de Sanger requería que cada comienzo de lectura fuera clonado para
producir un ADN de cadena simple. No obstante, con el desarrollo y mejora del
método de terminación de la cadena (ver más adelante), la secuenciación de
Maxam y Gilbert ha quedado en desuso debido a su complejidad técnica, el uso
extensivo de productos químicos peligrosos y dificultades para escalarla.
Además, a diferencia del método de terminación de la cadena, los reactivos que
se usan en el método de Maxam y Gilbert no se pueden adaptar para utilizarse en
un kit biológico estándar.
Secuenciación por terminador
fluorescente
 Una alternativa al marcado del cebador es el
marcado de los terminadores de la cadena, un método conocido como
"secuenciación por terminador fluorescente". La mayor ventaja de este
método es que la secuenciación se puede llevar a cabo en una sola reacción, en
lugar de en cuatro reacciones como en el método del cebador marcado. En una
secuenciación por terminador fluorescente se marcan cada uno de los cuatro
didesoxinucleótidos que terminan la cadena con un colorante fluorescente
diferente, con fluorescencias a diferentes longitudes de onda. Este método es
atractivo por su gran capacidad y rapidez y actualmente es el método de
referencia en la secuenciación automatizada con analizadores de secuencia
controlados por computadora (ver más abajo). Entre sus limitaciones potenciales
están los efectos de los terminadores fluorescentes en el fragmento de ADN, que
produce alturas y formas de picos desiguales en los registros de secuencia de
ADN del cromatograma tras la electroforesis capilar. Este problema
se ha solventado en gran medida con la introducción de nuevos sistemas
enzimáticos de polimerasas de ADN y colorantes que minimizan la variabilidad de
la incorporación, así como métodos para eliminar los "pegotes de
colorante" producidos por ciertas características químicas de los
colorantes que pueden dar lugar a artefactos en los registros de secuencia de
ADN. El método de secuenciado por terminador fluorescente junto con
analizadores de secuencia de ADN de alto rendimiento se utiliza ahora para la
inmensa mayoría de los proyectos de secuenciación, puesto que es más fácil de
llevar a cabo y tiene un coste menor que los anteriores métodos de
secuenciación.
Una alternativa al marcado del cebador es el
marcado de los terminadores de la cadena, un método conocido como
"secuenciación por terminador fluorescente". La mayor ventaja de este
método es que la secuenciación se puede llevar a cabo en una sola reacción, en
lugar de en cuatro reacciones como en el método del cebador marcado. En una
secuenciación por terminador fluorescente se marcan cada uno de los cuatro
didesoxinucleótidos que terminan la cadena con un colorante fluorescente
diferente, con fluorescencias a diferentes longitudes de onda. Este método es
atractivo por su gran capacidad y rapidez y actualmente es el método de
referencia en la secuenciación automatizada con analizadores de secuencia
controlados por computadora (ver más abajo). Entre sus limitaciones potenciales
están los efectos de los terminadores fluorescentes en el fragmento de ADN, que
produce alturas y formas de picos desiguales en los registros de secuencia de
ADN del cromatograma tras la electroforesis capilar. Este problema
se ha solventado en gran medida con la introducción de nuevos sistemas
enzimáticos de polimerasas de ADN y colorantes que minimizan la variabilidad de
la incorporación, así como métodos para eliminar los "pegotes de
colorante" producidos por ciertas características químicas de los
colorantes que pueden dar lugar a artefactos en los registros de secuencia de
ADN. El método de secuenciado por terminador fluorescente junto con
analizadores de secuencia de ADN de alto rendimiento se utiliza ahora para la
inmensa mayoría de los proyectos de secuenciación, puesto que es más fácil de
llevar a cabo y tiene un coste menor que los anteriores métodos de
secuenciación.
Secuenciación
alelo-específica por bisulfito
La secuenciación por bisulfito es una
variante de la secuenciación Sanger utilizada para el mapeo de metilaciones
alelo-específicas en los sitios CpG. Se extrae el ADN a secuenciar y se
fragmenta con el fin de facilitar la posterior desnaturalización a la que se va
a someter para separar las dos hebras de ADN. A continuación se incuba en
presencia de bisulfito entre 15 y 20 horas. Dicho compuesto actúa desaminando
las citosinas del ADN convirtiéndolas en uracilo. Sin embargo, es incapaz de
actuar sobre aquellas que se encuentren metiladas. El siguiente paso consiste
en la amplificación de la región que queremos mapear por PCR y su posterior
secuenciación. Finalmente, se compara la secuencia obtenida con la de un
control que no ha sido sometido a la acción del bisulfito: aquellas citosinas
que estuviesen metiladas aparecerán como tales en el control, mientras que en
la muestra tratada serán sustituidas por una timina.
La secuenciación alelo-específica por bisulfito
presenta algunas limitaciones, entre las cuales se encuentran las siguientes:
-Conversión incompleta: La secuenciación
mediante bisulfito depende de la conversión de cada uno de los residuos de citosina
no metilados a uracilo. Si la conversión es incompleta, los posteriores
análisis que se realicen interpretarán incorrectamente las citosinas no
metiladas no convertidas en uracilo como citosinas metiladas, dando lugar a
falsos positivos para la metilación.Sólo las citosinas que se encuentren en
cadenas simples de ADN serán susceptibles de ser atacadas por bisulfito por lo
que el paso de desnaturalización del ADN es crítico en este análisis.Es
importante por tanto optimizar parámetros como la temperatura y la
concentración de sales para mantener el ADN desnaturalizado y así permitir una
conversión completa por bisulfito. Por otra parte también se ha propuesto que
embebiendo el ADN en gel de agarosa se incrementa el grado de separación debido
a que se mantienen las cadenas separadas.
-Degradación del ADN durante el tratamiento con
bisulfito: Es una de las limitaciones más importantes de este método y
tiene lugar paralelamente a la conversión. Para que se produzca una total
conversión son necesarias una serie de condiciones tales como largos tiempos de
incubación, temperatura elevada y altas concentraciones de bisulfito. Estas
condiciones en su conjunto pueden promover la degradación del ADN en una
proporción elevada. Esto puede suponer un grave problema si partimos de
muestras con una baja concentración de ADN o de muestras tomadas post-mortem,
además dicha degradación dará lugar a pequeñas roturas al azar a lo largo de la
cadena de ADN lo que puede dar lugar a errores durante la amplificación por
PCR.
-Desulfonación incompleta de los residuos de
pirimidina: Una inadecuada alcalinización de la solución puede tener como
consecuencia una incompleta desulfonación de los residuos de pirimidina, lo
cual a su vez puede afectar negativamente a las polimerasas de ADN, las cuales
serán incapaces de replicar la cadena molde.Esta situación se puede evitar
monitorizando el pH de la solución asegurando así que se produzca una
desulfonación completa.
CONCLUSION
El ADN contiene la información hereditaria
correspondiente a la especie. Y el ARN requiere para la sintesis de
proteinas la presencia de los ribosomas en las células ya que en el momento de
la duplicación de los cromosomas la moléculas de ADN de abre gradualmente por
los puentes de hidrógeno. El papel de las moléculas de ADN en la transmisión
del codigo genético rompiendo células de Escherichia Coli, una bacteria
de la flora intestinal, separando sus componentes en varias fracciones.
Pero si el ADN es el responsable de la transmisión de la información genética
debe ser capaz, no solo de reproducirse, con lo cual se consigue conservar esta
información de padre a hijos sino también debe poder transmitirlo.
BIBLIOGRAFIA
http://www.ucm.es/info/genetica/grupod/Replicacion/Replicacion.htm
http://www.hiperbiologia.net/adn/adntema1.htm
http://www.biologia.arizona.edu/molecular_bio/problem_sets/nucleic_acids/06t.html
Libro: Biología, quinta edición. (Salomón Berg
Martín).
- Ansorge, W.J. (2009). «Next-generation DNA
sequencing techniques.». New Biotechnology 25 (4).
- Diggle, M. A. (2004). «Pyrosequencing™». Molecular
Biotechnology 28.
- França, L. T. C. (2002). «A review of DNA
sequencing techniques.». Quarterly Reviews of Biophysics 35 (2).
- Hutchinson, C. A. (2007). «sequencing: bench
to bedside and beyond.». Nucleic Acids Research 35 (18).
 El experimento de Meselson-Stahl fue un experimento realizado en 1957 por Matthew Meselson y Franklin Stahl en
el que se demostró que la replicación de ADN era semiconservadora.
Una replicación semiconservadora es aquella en que la cadena de dos filamentos
en hélice del ADN se replica de forma tal que cada una de las dos cadenas de ADN formadas consisten en un filamento
proveniente de la hélice original y un filamento nuevo sintetizado.
El experimento de Meselson-Stahl fue un experimento realizado en 1957 por Matthew Meselson y Franklin Stahl en
el que se demostró que la replicación de ADN era semiconservadora.
Una replicación semiconservadora es aquella en que la cadena de dos filamentos
en hélice del ADN se replica de forma tal que cada una de las dos cadenas de ADN formadas consisten en un filamento
proveniente de la hélice original y un filamento nuevo sintetizado.
 Kornberg (discípulo de Ochoa), en 1956,
aisló la enzima ADN-polimerasa (enzima capaz de unir nucleótidos de ADN in
vitro), realizó la siguiente experiencia:
Kornberg (discípulo de Ochoa), en 1956,
aisló la enzima ADN-polimerasa (enzima capaz de unir nucleótidos de ADN in
vitro), realizó la siguiente experiencia: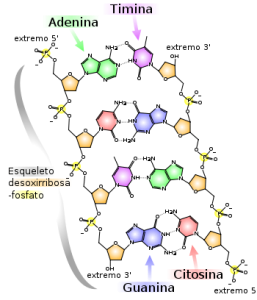 Una secuencia de ADN o secuencia genética es una sucesión de
letras representando la estructura primaria de una molécula real o hipotética de ADN o banda,
con la capacidad de transportar información.
Una secuencia de ADN o secuencia genética es una sucesión de
letras representando la estructura primaria de una molécula real o hipotética de ADN o banda,
con la capacidad de transportar información. En 1976-1977, Allan Maxam y Walter Gilbert desarrollaron un método para secuenciar ADN basado
en la modificación química del ADN y posterior escisión en bases específicas
Aunque Maxam y Gilber publicaron su secuenciación química dos años antes del
trascendental artículo de Sanger y Coulson sobre su método de secuenciación
"más-menos",la secuenciación de Maxam y Gilbert rápidamente se hizo
más popular hasta que se pudo utilizar ADN directamente, mientras que el método
inicial de Sanger requería que cada comienzo de lectura fuera clonado para
producir un ADN de cadena simple. No obstante, con el desarrollo y mejora del
método de terminación de la cadena (ver más adelante), la secuenciación de
Maxam y Gilbert ha quedado en desuso debido a su complejidad técnica, el uso
extensivo de productos químicos peligrosos y dificultades para escalarla.
Además, a diferencia del método de terminación de la cadena, los reactivos que
se usan en el método de Maxam y Gilbert no se pueden adaptar para utilizarse en
un kit biológico estándar.
En 1976-1977, Allan Maxam y Walter Gilbert desarrollaron un método para secuenciar ADN basado
en la modificación química del ADN y posterior escisión en bases específicas
Aunque Maxam y Gilber publicaron su secuenciación química dos años antes del
trascendental artículo de Sanger y Coulson sobre su método de secuenciación
"más-menos",la secuenciación de Maxam y Gilbert rápidamente se hizo
más popular hasta que se pudo utilizar ADN directamente, mientras que el método
inicial de Sanger requería que cada comienzo de lectura fuera clonado para
producir un ADN de cadena simple. No obstante, con el desarrollo y mejora del
método de terminación de la cadena (ver más adelante), la secuenciación de
Maxam y Gilbert ha quedado en desuso debido a su complejidad técnica, el uso
extensivo de productos químicos peligrosos y dificultades para escalarla.
Además, a diferencia del método de terminación de la cadena, los reactivos que
se usan en el método de Maxam y Gilbert no se pueden adaptar para utilizarse en
un kit biológico estándar. Una alternativa al marcado del cebador es el
marcado de los terminadores de la cadena, un método conocido como
"secuenciación por terminador fluorescente". La mayor ventaja de este
método es que la secuenciación se puede llevar a cabo en una sola reacción, en
lugar de en cuatro reacciones como en el método del cebador marcado. En una
secuenciación por terminador fluorescente se marcan cada uno de los cuatro
didesoxinucleótidos que terminan la cadena con un colorante fluorescente
diferente, con fluorescencias a diferentes longitudes de onda. Este método es
atractivo por su gran capacidad y rapidez y actualmente es el método de
referencia en la secuenciación automatizada con analizadores de secuencia
controlados por computadora (ver más abajo). Entre sus limitaciones potenciales
están los efectos de los terminadores fluorescentes en el fragmento de ADN, que
produce alturas y formas de picos desiguales en los registros de secuencia de
ADN del cromatograma tras la electroforesis capilar. Este problema
se ha solventado en gran medida con la introducción de nuevos sistemas
enzimáticos de polimerasas de ADN y colorantes que minimizan la variabilidad de
la incorporación, así como métodos para eliminar los "pegotes de
colorante" producidos por ciertas características químicas de los
colorantes que pueden dar lugar a artefactos en los registros de secuencia de
ADN. El método de secuenciado por terminador fluorescente junto con
analizadores de secuencia de ADN de alto rendimiento se utiliza ahora para la
inmensa mayoría de los proyectos de secuenciación, puesto que es más fácil de
llevar a cabo y tiene un coste menor que los anteriores métodos de
secuenciación.
Una alternativa al marcado del cebador es el
marcado de los terminadores de la cadena, un método conocido como
"secuenciación por terminador fluorescente". La mayor ventaja de este
método es que la secuenciación se puede llevar a cabo en una sola reacción, en
lugar de en cuatro reacciones como en el método del cebador marcado. En una
secuenciación por terminador fluorescente se marcan cada uno de los cuatro
didesoxinucleótidos que terminan la cadena con un colorante fluorescente
diferente, con fluorescencias a diferentes longitudes de onda. Este método es
atractivo por su gran capacidad y rapidez y actualmente es el método de
referencia en la secuenciación automatizada con analizadores de secuencia
controlados por computadora (ver más abajo). Entre sus limitaciones potenciales
están los efectos de los terminadores fluorescentes en el fragmento de ADN, que
produce alturas y formas de picos desiguales en los registros de secuencia de
ADN del cromatograma tras la electroforesis capilar. Este problema
se ha solventado en gran medida con la introducción de nuevos sistemas
enzimáticos de polimerasas de ADN y colorantes que minimizan la variabilidad de
la incorporación, así como métodos para eliminar los "pegotes de
colorante" producidos por ciertas características químicas de los
colorantes que pueden dar lugar a artefactos en los registros de secuencia de
ADN. El método de secuenciado por terminador fluorescente junto con
analizadores de secuencia de ADN de alto rendimiento se utiliza ahora para la
inmensa mayoría de los proyectos de secuenciación, puesto que es más fácil de
llevar a cabo y tiene un coste menor que los anteriores métodos de
secuenciación.